
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
1
Protein Synthesis
and drugs that inhibit
protein synthesis
Objectives:
1. To understand the steps involved in the
translation process that leads to protein synthesis
2. To understand and know about all the structures
and molecules involved in protein synthesis
3. know about the drugs that inhibits protein
synthesis and the mechanism of inhibition
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
2
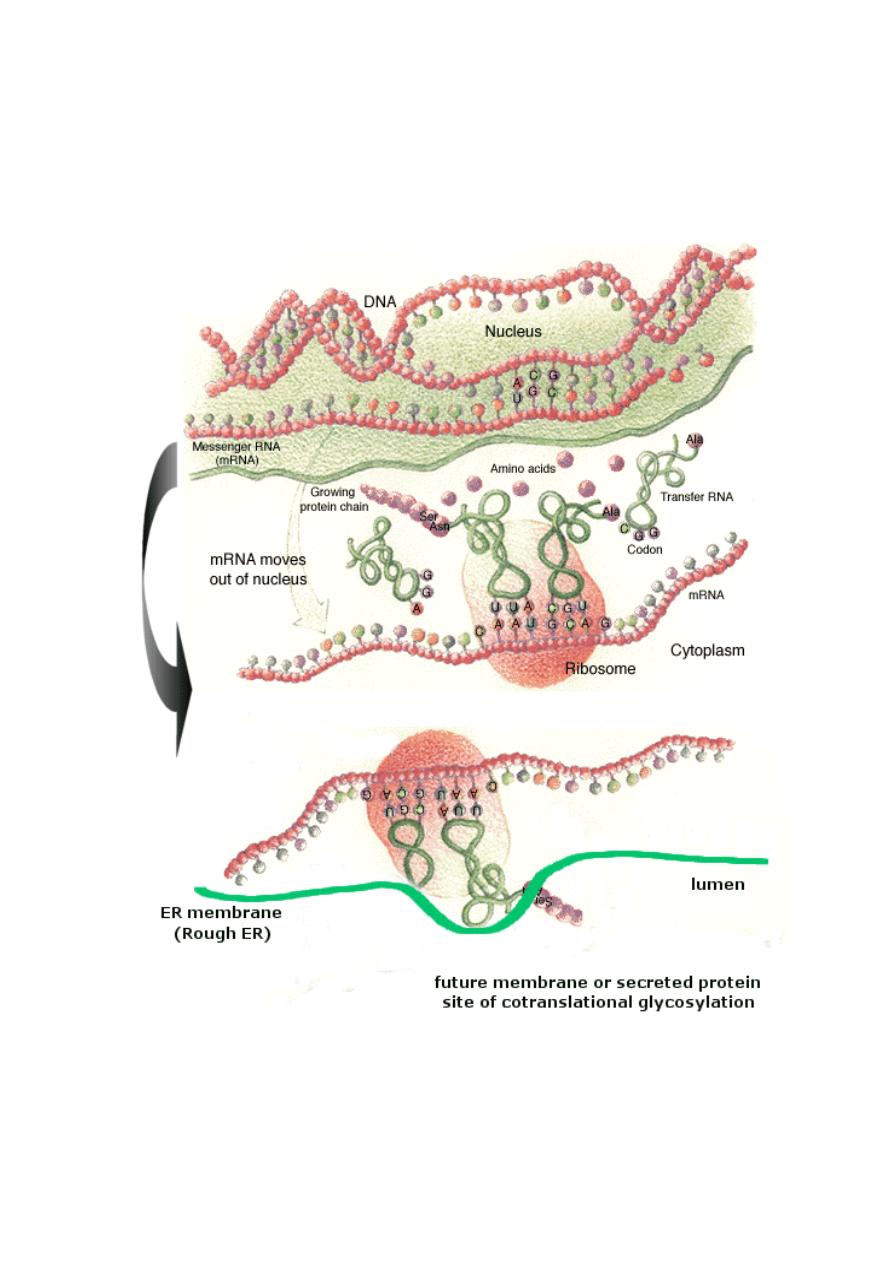
Protein biosynthesis is the process in which cells build proteins. The term
is sometimes used to refer only to protein translation, but more often it
refers to a multi-step process, beginning with transcription and ending with
proteintranslation.
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
3
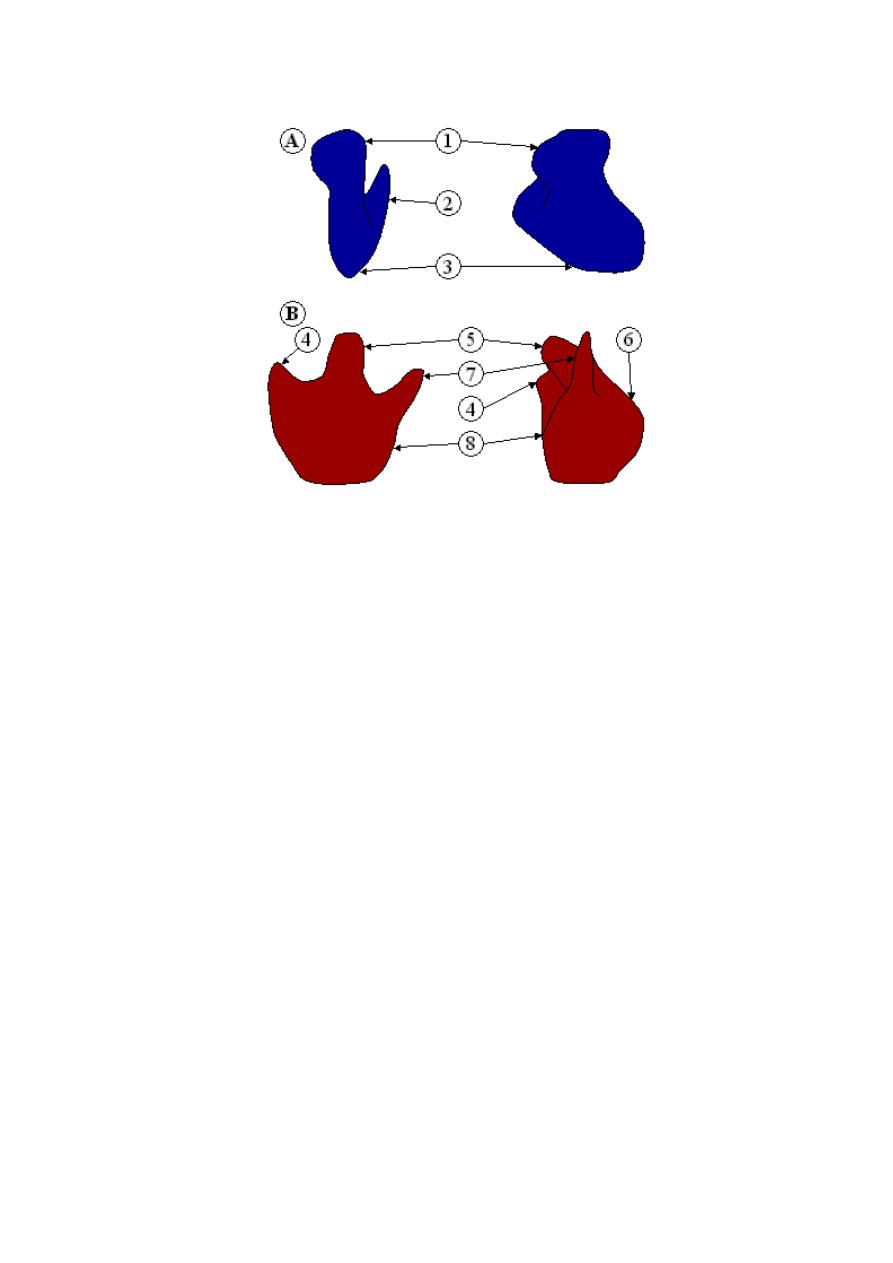
Ribosome
Figure: Ribosome structure indicating small subunit (A) and large subunit
(B). Side and front view.(1) Head. (2) Platform. (3) Base. (4) Ridge. (5)
Central protuberance. (6) Back. (7) Stalk. (8) Front.
A ribosome is an organelle composed of rRNA (synthesized in the
nucleolus) and ribosomal proteins. It translates mRNA into a polypeptide
chain (e.g., a protein). It can be thought of as a factory that builds a protein
from a set of genetic instructions.
Free ribosomes
Free ribosomes occur in all cells. Free ribosomes usually produce proteins
that are used in the cytosol or in the organelle they occur in.
Membrane bound ribosomes
When certain proteins are synthesized by a ribosome, it can become
"membrane-bound", associated with the membrane of the nucleus and the
rough endoplasmic reticulum (in eukaryotes only) for the time of synthesis.
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
4
The ribosomal subunits of prokaryotes and eukaryotes are quite similar.
However, prokaryotes use 70S ribosomes, each consisting of a (small) 30S
and a (large) 50S subunit, whereas eukaryotes use 80S ribosomes, each
consisting of a (small) 40S and a bound (large) 60S subunit.[
The unit S
means Svedberg units, a measure of the rate of sedimentation of a particle in a
centrifuge, where the sedimentation rate is associated with the size of the particle
].
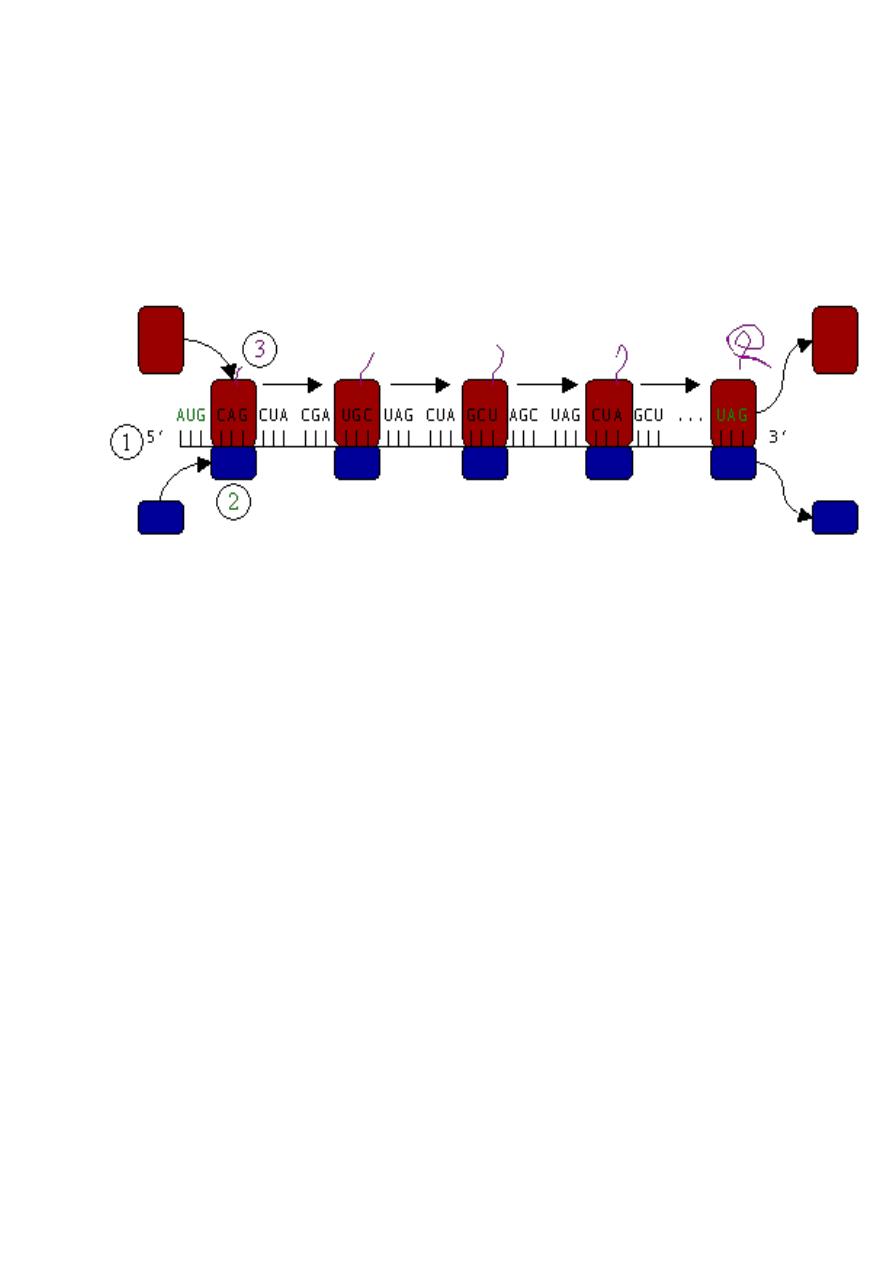
Figure : Translation (1) of mRNA by a ribosome (2) into a polypeptide
chain (3). The mRNA begins with a start codon (AUG) and ends with a stop
codon (UAG).
Translation (also called protein biosynthesis or polypeptide synthesis)
is the second process in gene expression. In translation, messenger RNA
is used as a template to produce a specific polypeptide according to the
rules specified by the genetic code.
Phases
Translation proceeds in three phases: initiation, elongation, and termination
(all describing the growth of the amino acid chain, or polypeptide that is the
product of translation).

Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
5
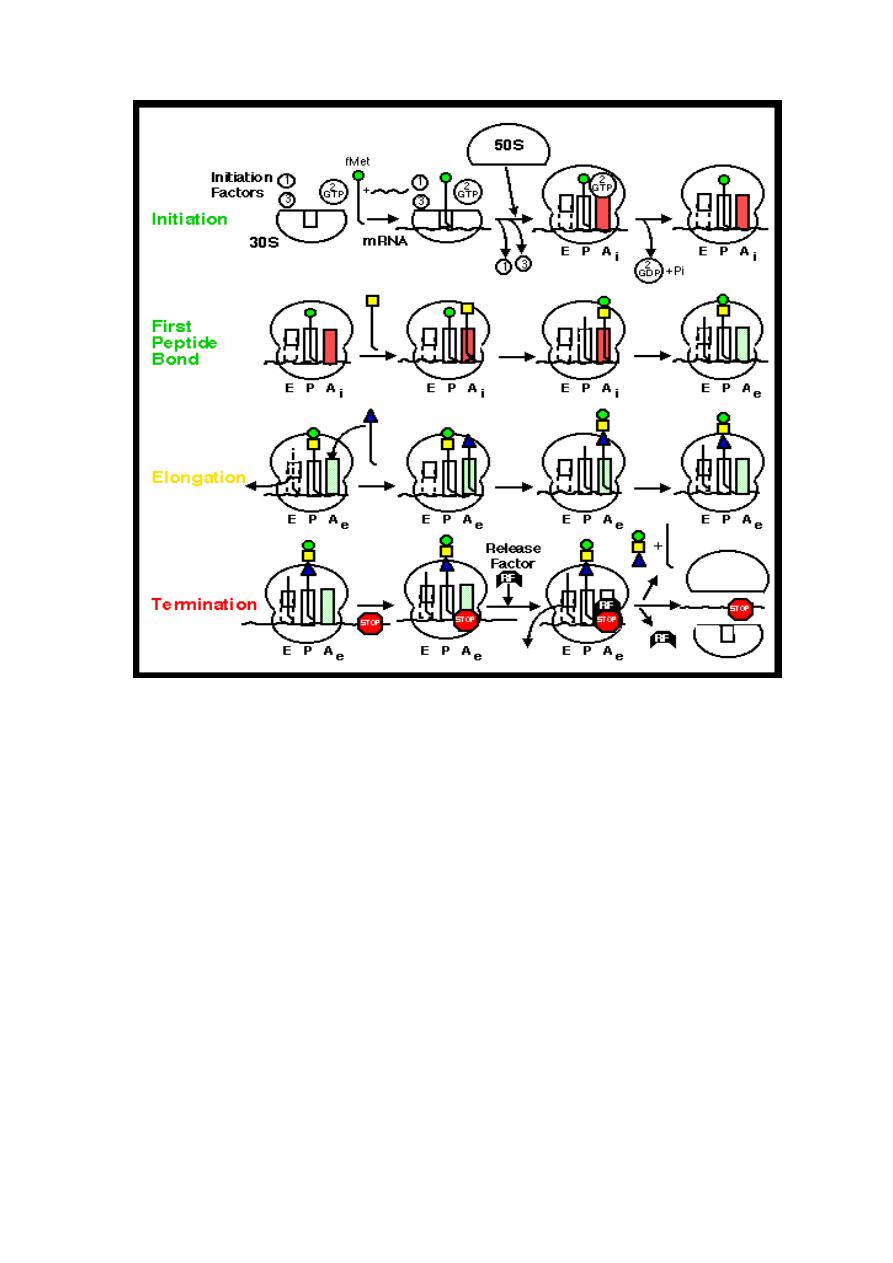
1. Initiation of translation involves the small ribosomal subunit binding
to the 'start' codon on the mRNA, which indicates where the mRNA
starts coding for the protein. This codon is most commonly an AUG.
In eukaryotes amino acid encoded by the start codon is methionine.
In bacteria, the protein starts instead with the modified amino acid N-
formyl methionine (f-Met). In f-Met, the amino group has been
blocked by a formyl group to form an amide, so this amino group can
not form a peptide bond. This is not a problem because the f-Met is at
the amino terminus of the protein.
2. The large subunit then forms a complex with the small subunit, and
elongation proceeds. A new activated tRNA enters the A site of the
ribosome and base pairs with the mRNA. The enzyme peptidyl
transferase forms a peptide bond between the adjacent amino acids.
As this happens, the amino acid on the P site leaves its tRNA and
joins the tRNA at the A site. The ribosome then moves in relation to
the mRNA shifting the tRNA at the A site on to the P whilst releasing
the empty tRNA, this process is known as translocation.
3. This procedure repeats until the ribosome encounters one of three
possible stop codons, where translation is terminated. This stalls
protein growth, and release factors, proteins which mimic tRNA, enter
the A site and release the protein in to the cytoplasm.
Synthesis of proteins can take place extremely quickly. This is aided by
multiple ribosomes being able to attach themselves to one mRNA chain,
thus allowing multiple proteins to be constructed at once. An mRNA chain
with multiple ribosomes is called a polysome. Also, as prokaryotes have no
nucleus, an mRNA can be translated while it is still being transcribed. This
is not possible in eukaryotes as translation occurs in the cytoplasm,
whereas transcription occurs in the nucleus.
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
6
Protein Synthesis in Eukaryotes
A major difference between eukaryotes and prokaryotes is that, in a
typical eukaryotic cell, protein synthesis takes place in the cytoplasm while
transcription and RNA processing take place in the nucleus. In bacteria,
these two processes can be coupled so that protein synthesis can start
even before transcription has finished.
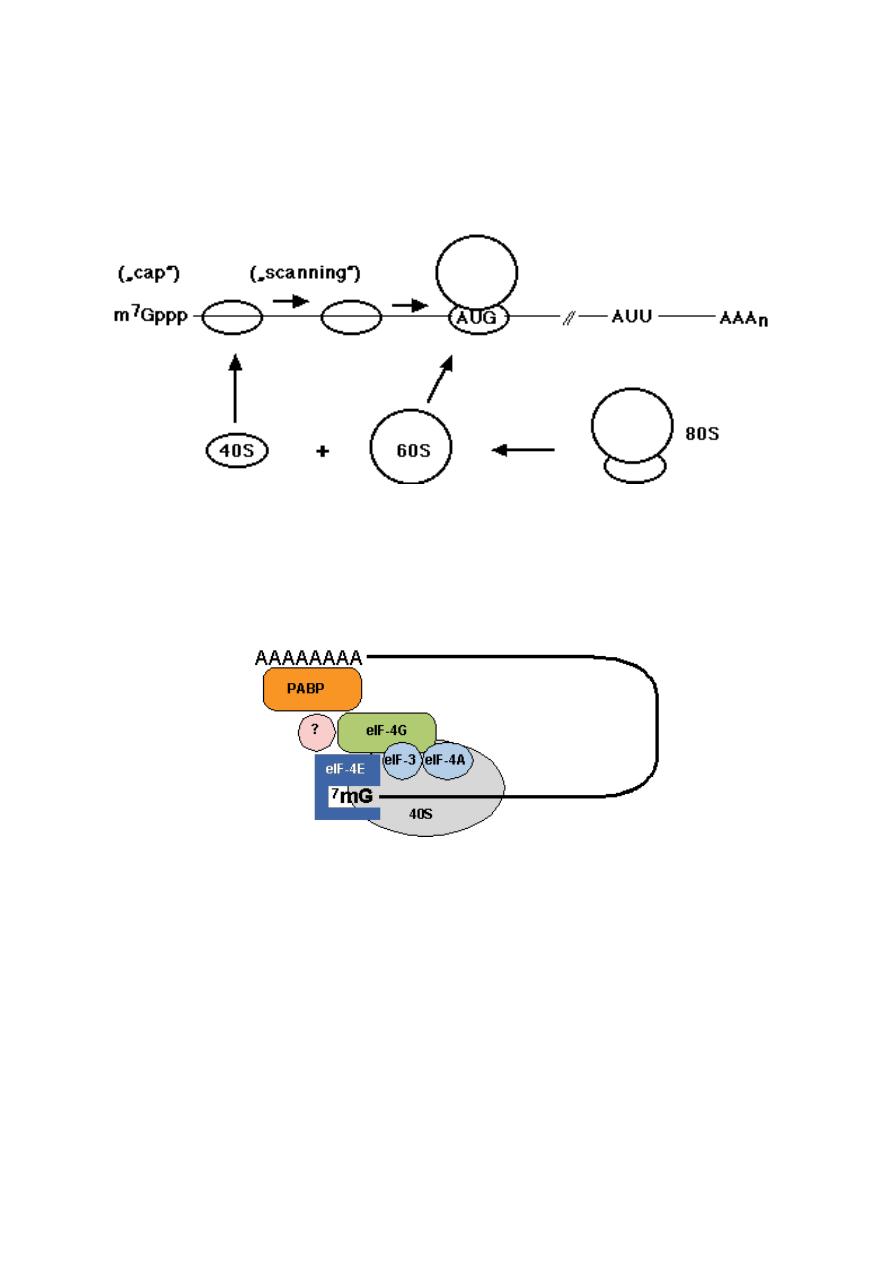
INITIATION
The cap-dependent translation initiation pathway
Cap-dependent initiation is the major translation initiation pathway in
eukaryotes
eukaryotic mRNAs are monocistronic, capped at the 5' end and
polyadenylated at the 3' end
ribosomes dissociate into 40S and 60S subunits
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
7
40S subunits locate the initiator AUG codon by scanning the mRNA
from the cap structure in the 3' direction for the first AUG codon
at the AUG codon the 60S ribosomal subunit joins the 40S initiation
complex to form an 80S ribosome competent for translation
elongation:
Fig.: Principle of cap-dependent translation initiation. AUU, stop codon;
AAAn, poly(A) tract.
A large number of proteins, the eukaryotic translation initiation factors (eIF)
catalyze individual steps in the pathway.
ELONGATION: The elongation in eukaryotes is very similar to that in
prokaryotes.
TERMINATION: Mechanism in eukaryotes is similar to that in prokaryotes
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
8
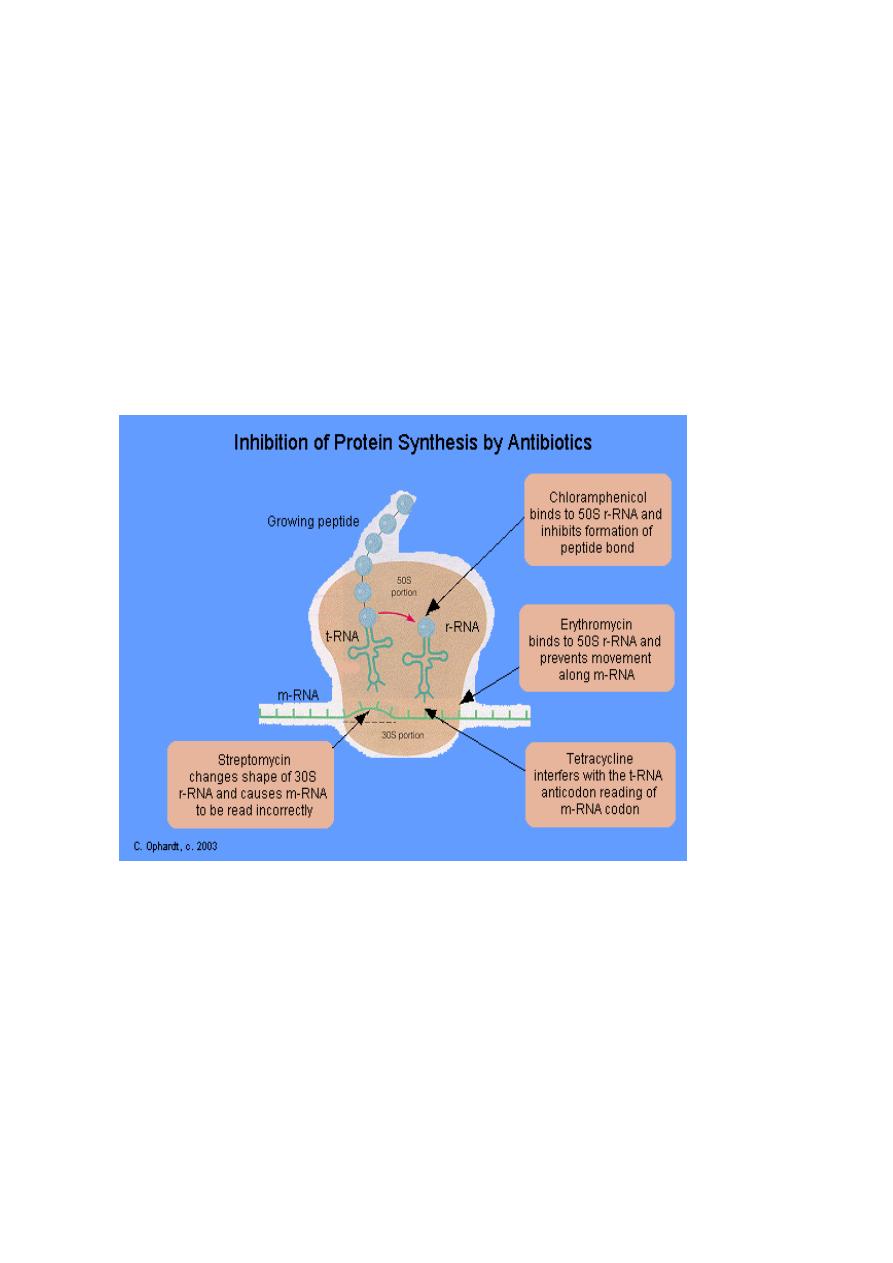
Drugs that inhibits protein synthesis
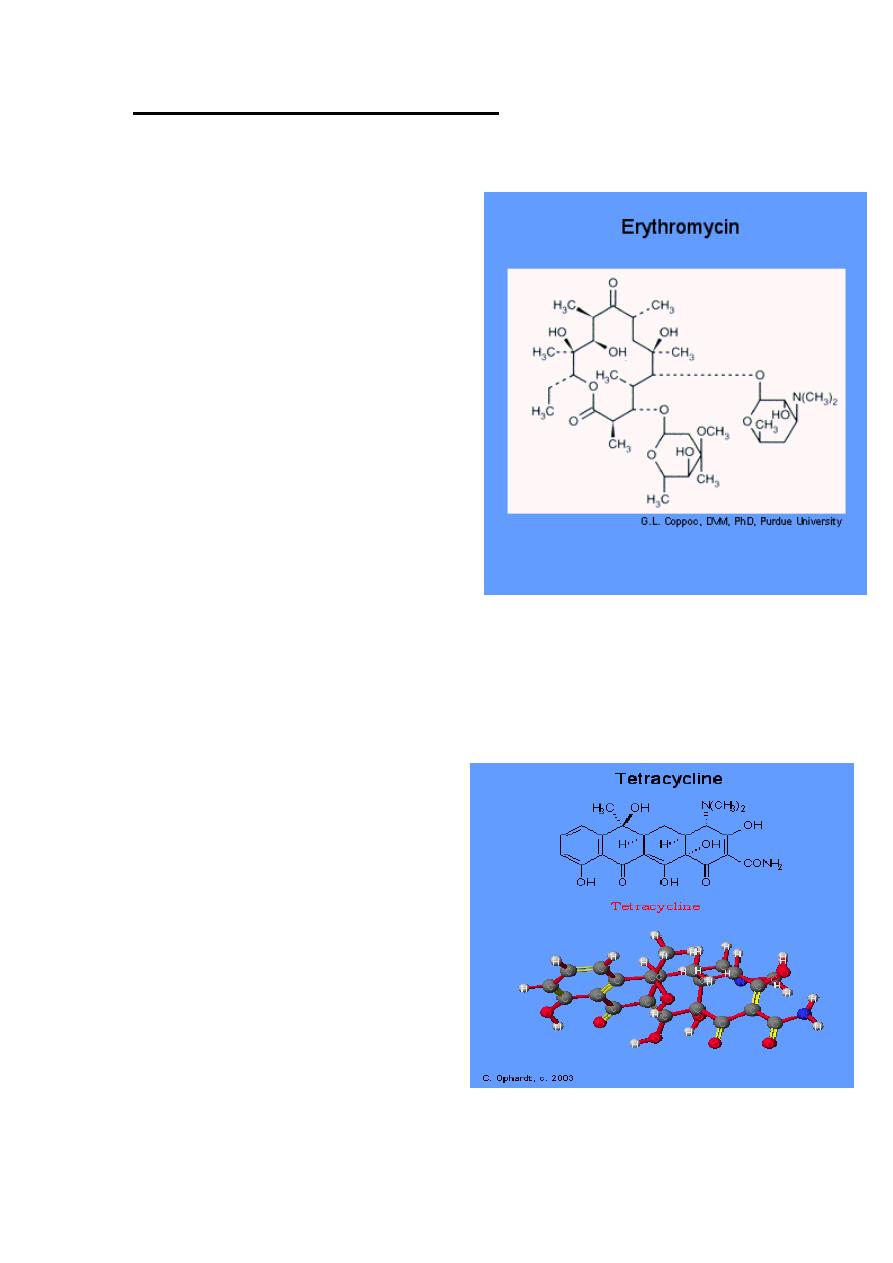
1. Erythromycin
Mechanism of Action
Erythromycin inhibit protein synthesis
by binding to the 23S rRNA molecule
(in the 50S subunit) of the bacterial
ribosome blocking the exit of the
growing peptide chain. (Humans do
not have 50 S ribosomal subunits, but
have ribosomes composed of 40 S and
60 S subunits). Certain resistant
microorganisms with mutational
changes in components of this subunit
of the ribosome fail to bind the drug.
The association between erythromycin
and the ribosome is reversible and
takes place only when the 50 S subunit
is free from tRNA molecules bearing
nascent peptide chains. The non
ionized from of the drug is
considerably more permeable to cells, and this probably explains the
increased antimicrobial activity that is observed in alkaline pH.
2. Tetracyclines:
Tetracyclines have the broadest
spectrum of antimicrobial activity.
Four fused 6-membered rings, as
shown in the figure below, form the
basic structure from which the
various tetracyclines are made
Mechanism of Action:
Tetracyclines inhibit bacterial protein
synthesis by blocking the attachment
of the transfer RNA-amino acid to the
ribosome. More precisely they are
inhibitors of the codon-anticodon
interaction. Tetracyclines can also
inhibit protein synthesis in the host,
but are less likely to reach the concentration required because eukaryotic
cells do not have a tetracycline uptake mechanism.
Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
9
3. Streptomycin: Streptomycin binds to the 30S ribosome and changes its
shape so that it and inhibits protein synthesis by causing a
misreading of messenger RNA information.
4. Chloramphenicol:
Chloromycetin is also a broad spectrum antibiotic that possesses activity
similar to the tetracylines. At present, it is the only antibiotic prepared
synthetically. It is reserved for treatment of serious infections because it is
potentially highly toxic to bone marrow cells. It inhibits protein synthesis by
attaching to the ribosome and interferes with the formation of peptide
bonds between amino acids.

Lecture 4: Protein synthesis and translation in prokaryotic and eukaryotic cells and drugs that
inhibit this process
2014
Prof.Dr. H.D.El-Yassin
11
Conclusion:
1. Proteins are produced by the process of translation which involves three main
steps:
a. Initiation involves formation of a complex containing the initial methionyl-
tR
NA bound to the AUG “start” codon of the mRNA and to the “P” site of
the ribosome.
b. Elongation of the polypeptide involves three steps:
i. binding of an aminoacyl-
tRNA to the “A” site on the ribosome where
it base-pairs with the secondcodon on the mRNA;
ii. formation of a peptide bond between the first and second amino
acids; and
iii. (c) translocation, movement of the mRNA relative to the ribosome,
so that the third mRNA codon moves into the “A” site.
c. These three elongation steps are repeated until a termination codon
aligns with the site on the ribosome where the next aminoacyl-tRNA would
normally bind.
2. Protein synthesis occurs on ribosomes and is directed by mRNA. mRNA with
multiple ribosomes is known as a polysome.
3. each step in the protein synthesis might me inhibited by a certain drug
