
2
Lec.1 DNA STRUCTURE & SYNTHESIS
Nucleic acids are required for the storage and expression of
genetic information. There are two chemically distinct types of
nucleic acids: deoxyribonucleic acid (DNA) and ribonucleic
acid DNA, the storehouse of genetic information
The genetic information found in DNA is copied and
transmitted to daughter cells through DNA replication. The
DNA contained in a fertilized egg encodes the information
that directs the development of an organism. This
development may involve the production of billions of cells.
Each cell is specialized, expressing only those functions that
are required for it to perform its role in maintaining the
organism. Therefore, DNA must be able to not only replicate
precisely each time a cell divides, but also to have the
information that it contains be selectively expressed.
Transcription (RNA synthesis) is the first stage in the
expression of genetic information.
Next, the code contained in the nucleotide sequence of
messenger RNA molecules is translated, thus completing gene
expression. This flow of information from DNA to RNA to
protein is termed the "central dogma of molecular biology"
The eukaryotic cell cycle
The events surrounding DNA replication and cell division
(mitosis) are coordinated to produce the cell cycle. The period
preceding replication is called the G1 phase (Gap1).
DNA replication occurs during the S (synthesis) phase.
Following DNA synthesis, there is another period (G2 phase,
Gap2) before mitosis (M). Cells that have stopped dividing,
such as mature neurons, are said to have gone out of the cell
cycle into the GO phase.
STRUCTURE OF DNA
DNA is a polydeoxyribonucleotides that contains many
nucleotides covalently linked by 3-5 phosphodiester bonds.
DNA exists as a double-stranded molecule, in which the two
strands wind around each other, forming a double helix.
Nucleotides are composed of a nitrogenous base, a pentose
monosaccharide, and one, two, or three phosphate groups. The
nitrogen-containing bases belong to two families of
compounds: the purines and the pyrimidines.
Both DNA and RNA contain the same purine bases: adenine
(A) and guanine (G). Both DNA and RNA contain the
pyrimidine cytosine (C), but they differ in their second
pyrimidine base: DNA contains thymine (T), whereas RNA
contains uracil (U). DNA is found associated with various
types of proteins (known collectively as nucleoprotein,
present in the nucleus,
3-5phosphodiester bonds
3-5 phosphodiester bonds bonds join the 5'-hydroxyl group of
the deoxypentose of one nucleotide to the 3'-hydroxyl group
of the deoxypentose of an adjacent nucleotide through a
phosphate group
Double helix
The two chains of DNA are coiled around a common axis
called the axis of symmetry. The chains are paired in an
antiparallel manner, that is, the 5'-end of one strand is paired
with the3'-end of the other strand
The spatial relationship between the two strands in the helix
creates a major (wide) groove and a minor (narrow) groove.
These grooves provide access for the binding of regulatory
proteins to their specific recognition sequences along the DNA
chain. Certain anticancer drugs, such as actinomycin D), exert
their cytotoxic effect by intercalating into the narrow groove
of the DNA double helix, thus interfering with RNA and DNA
synthesis.
Base pairing:
The bases of one strand of DNA are paired with the bases of
the second strand, so that an adenine is always paired with a
thymine and a cytosine is always paired with a guanine
Therefore, one polynucleotide chain of the DNA double helix
is always the complement of the other. Given the sequence of
bases on one chain, the sequence of bases on the
complementary chain can be determined .in any sample of
double-stranded DNA, the amount of adenine equals the
amount of thymine, the amount of guanine equals the amount
of cytosine, and the total amount of purines equals the total
amount of pyrimidines .
The base pairs are held together by hydrogen bonds:
two hydrogen bonds between A and T and
three hydrogen bonds between G and C.These hydrogen
bonds, plus the hydrophobic interactions between the stacked
bases, stabilize the structure of the double helix.
Separation of the two DNA strands in the double helix;
The two strands of the double helix separate when hydrogen
bonds between the paired bases are disrupted.
A SYNTHESIS:
When the two strands of the DNA double helix are separated,
each can serve as a template for the replication of a new
complementary strand. This produces two daughter molecules,
each of which contains two DNA strands with an antiparallel
orientation. This process is called Semiconserative
replication because, although the parental duplex is separated
into two halves (and, therefore, is not "conserved" as an
entity), each of the individual parental strands remains intact
in one of the two new duplexes.

3
The enzymes involved in the DNA replication process are
template-directed polymerases that can synthesize the
complementary sequence of each strand with extraordinary
fidelity.
A. Separation of the two complementary DNA strands
In order for the two strands of the parental double helical
DNA to be replicated, they must first separate at least in a
small region, because the polymerases use only single-
stranded DNA as a template.
Replication begins at multiple sites along the DNA helix
(These sites include a short sequence composed almost
exclusively of AT base pairs. [This is referred to as a
consensus sequence, because the order of nucleotides is
essentially the same at each site.] Having multiple origins of
replication provides a mechanism for rapidly replicating the
great length of the DNA molecules
B.
Formation of the replication fork
As the two strands unwind and separate they form a
"V" where active synthesis occurs. This region is called the
replication fork.it moves along the DNA molecule as
synthesis occurs. Replication of double-stranded DNA is
bidirectional that is, the replication forks move in both
directions away from the origin.
1) Proteins required for DNA strand separation:
Initiation of DNA replication requires the recognition of the
origin of replication and/or the replication fork by a group of
proteins . These proteins are responsible for maintaining the
separation of the parental strands, and for unwinding the
double helix ahead of the advancing replication fork. These
proteins include the following:
a) DnaA protein:
Twenty to fifty monomers of dnaA protein bind to specific
nucleotide sequences at the origin of replication, which is
particularly rich in AT base pairs. This process causes the
double-stranded DNA to melt and the strands separate,
forming localized regions of single-stranded DNA.
b) Single-stranded DNA-binding (SSB) proteins:
Also called helix-destabilizing proteins, these bind only
to single-stranded DNA. These proteins have many
functions ; they keep the two strands of DNA separated in
the area of the replication origin, thus providing the single-
stranded template required by polymerases, also they
protect DNA from nucleases that cleave single-stranded
DNA.
c) DNA helicases: These enzymes bind to single-stranded
DNA near the replication fork, and then move into the
neighboring double-stranded region, forcing the strands
apart, unwinding the double helix. Helicases require
energy provided by ATP. When the strands separate, SSB
proteins bind, preventing reformation of the double helix.
Figure 29.16 Elongation of the leading and lagging strands.
2) Solving the problem of supercoils:
As the two strands of the double helix are separated, a
problem is encountered ,which is the appearance of positive
supercoils (also called supertwists) in the region of DNA
ahead of the replication fork. The accumulating positive
supercoils interfere with further unwinding of the double
helix, to solve this problem, there is a group of enzymes called
DNA which are responsible for removing supercoils in the
helix.
Type I DNA topoisomerases reversibly cut a single strand of
the double helix. They have both nuclease (strand-cutting)
and ligase (strand-resealing) activities.
Type II DNA topoisomerases bind tightly to the DNA double
helix and make transient breaks in both strands. The enzyme
then causes a second stretch of the DNA double helix to pass
through the break and, finally, reseals the break
C. Direction of DNA replication
1) Leading strand: The strand that is being copied in the
direction of the advancing replication fork is called the leading
strand and is synthesized almost continuously.
2) Lagging strand: The strand that is being copied in the
direction away from the replication fork is synthesized
discontinuously, with small fragments of DNA being copied
near the replication fork. These short stretches of
discontinuous DNA, termed okazaki fragments, are
eventually joined to become a single,continuous strand. The
new strand of DNA produced by this mechanism is termed the
lagging strand
D. RNA primer
DNA polymerases cannot initiate synthesis of a
complementary strand of DNA on a totally single-stranded
template. Rather, they require an RNA primer which is a
short, double-stranded region consisting of RNA base-paired
to the DNA template.

4
1) Primase: A specific RNA polymerase, called Primase
synthesizes the short stretches of RNA (approximately ten
nucleotides long) that are complementary and antiparalle to
the DNA template. in the resulting hybrid duplex, the U in
RNA pairs with A in DNA. These short RNA sequences are
constantly being synthesized at the replication fork on the
lagging strand, but only one RNA sequence at the origin of
replication required on the leading strand.
2) primosome Prior to the beginning of RNA primer synthesis on
the lagging strand, a complex of several proteins assembled
and binds to the single strand of DNA, displacing some of the
single-stranded DNA-binding proteins. This protein complex,
plus primase is called the primosome. initiates Okazaki
fragment formation by moving along the template for the
lagging strand , periodically recognizing specific sequences of
nucleotides that direct it to create an RNA primer.
E. Chain elongation
DNA polymerases elongate a new DNA strand by adding
deoxyribonucleotides, one at a time, to the 3'-end of the
growing. The sequence of nucleotides that are addeds dictated
by the base sequence of the template strand with which the
incoming nucleotides are paired.
1) DNA polymerase
DNA chain elongations catalyzed by Using the 3'-hydroxy
group of the RNA primer as the acceptor of the first
deoxyribonucleotide, DNA polymerase begins to add
nucleotides along the single-stranded template that specifies
the sequence of bases in the newly synthesized chain.
2) Proofreading of newly synthesized DNA:
Is highly important for the survival of an organism that the
nucleotide sequence of DNA be replicated with as few errors
as possible. Misreading of the template sequence could result
in perhaps lethal mutations. To ensure replication fidelity,
DNA polymerase has, in addition to its polymerase activity, a
"proofreading" activity, As each nucleotide is added to the
chain, DNA polymerase checks to make certain the added
nucleotide is, in fact, correctly matched to its complementary
base on the template. IF it is not, the exonuclease activity edits
the mistake.
F. Excision of RNA primers and their
replacement by DNA
DNA polymerase continues to synthesize DNA on the
lagging strand until it is blocked by proximity to an RNA
primer. RNA primers are removed by RNASE H and the gap
filled by DNA polymerase I
G. DNA ligase
The final phosphodiester linkage between the 5'-phosphate
group on the DNA chain and the 3'-hydroxyl group on the
chain is catalyzed by DNA ligase
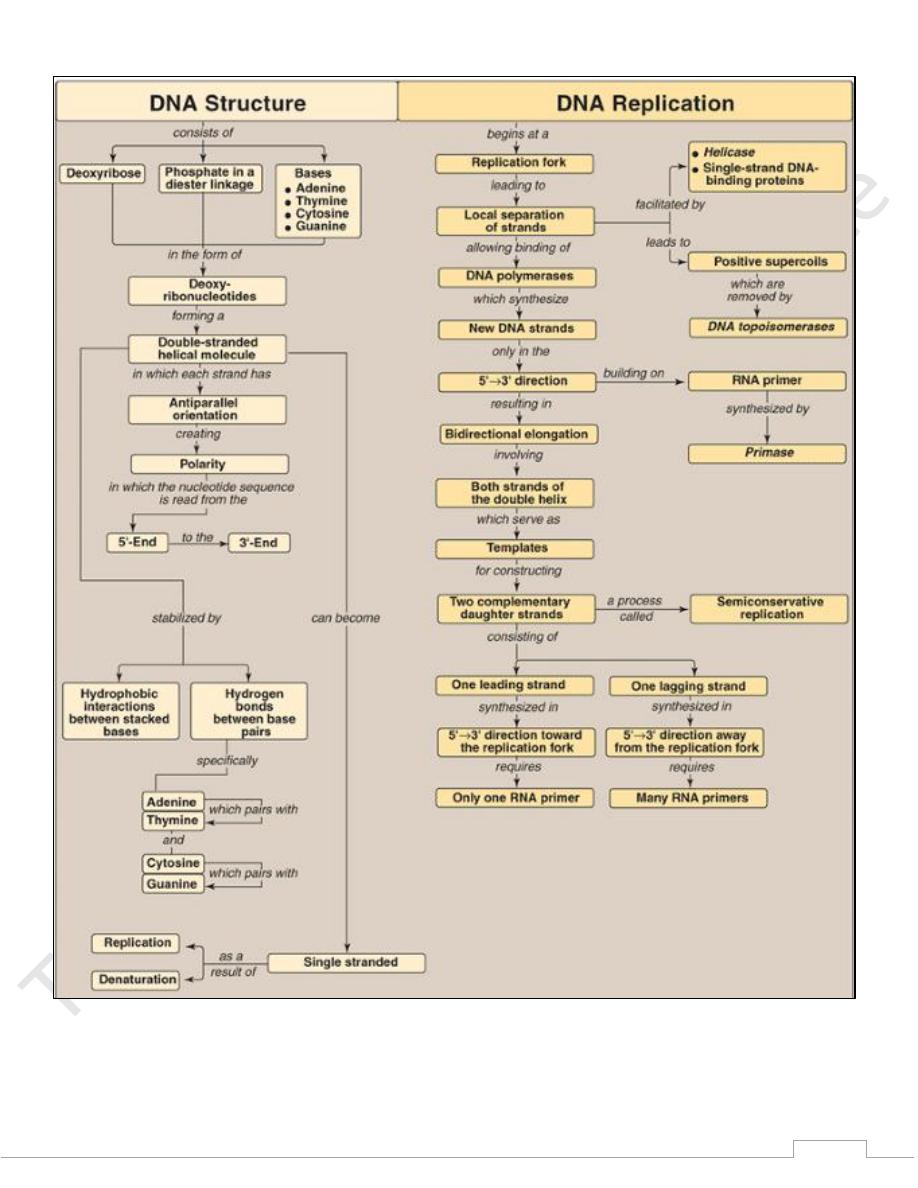
5
Figure 29.31 Key concept map for DNA structure,
replication, and repair. NHEJ = nonhomologous end-
joining; HR = homologous recombination.
